-
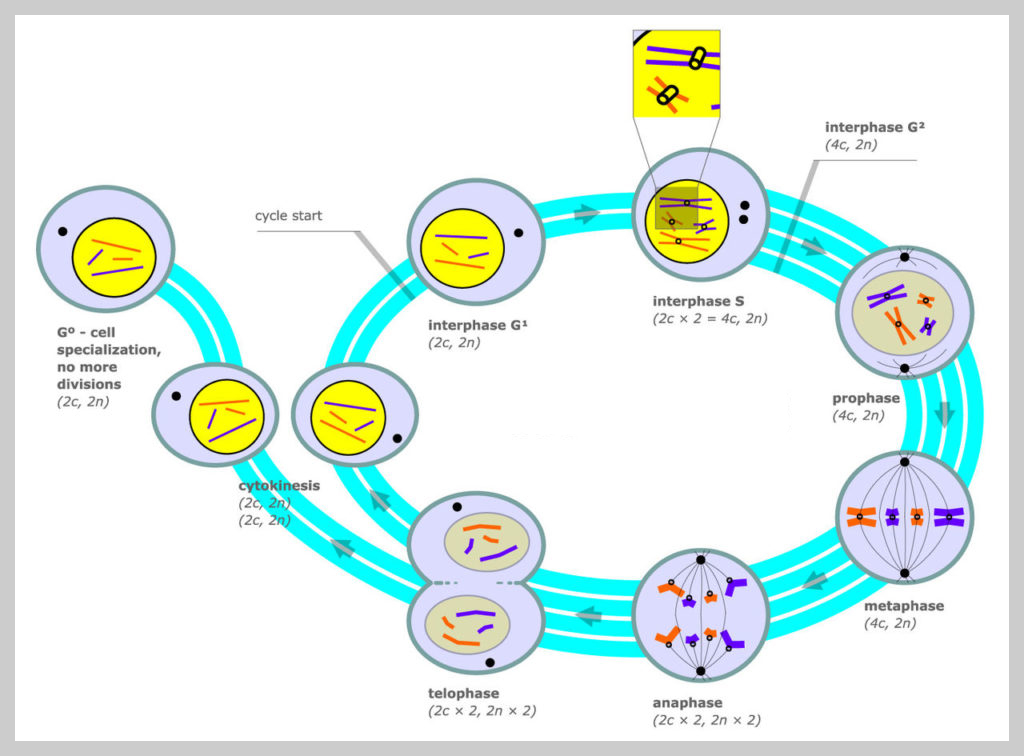
세포주기 (Cell cycle)란
DNA가 두 배가 되는 S기, 염색체 분할과 세포 분열이 일어나는 M기, 그리고 이 두 시기를 준비하는 중간 기간인 G1기와 G2기로 나누어집니다. 세포 증식은 G1, S, G2 및 M 단계의 순서가 주기적으로 반복됩니다.
출처: https://courses.lumenlearning.com/suny-wmopen-biology1/chapter/the-cell-cycle/
The Cell Cycle | Biology for Majors I
Identify the stages of the cell cycle, by picture and by description of major milestones The cell cycle is an ordered series of events involving cell growth and cell division that produces two new daughter cells. Cells on the path to cell division proceed
courses.lumenlearning.com
세포주기 측정 및 원리
세포주기의 각 단계에서 세포의 비율을 분석하기 위해 DNA 염색의 FACS 분석을 통해 이를 확인하고자 합니다. DNA 염색에 사용되는 PI 시약의 경우 PI (propidium iodide) 염색은 세포생존율이나 세포주기 분석 시 DNA 염색에 사용할 수 있는 시약입니다.
염색시약은 건강하고 살아있는 세포의 막과 핵막을 침투할 수 없으므로 염색 전 주로 에탄올이나 파라포름알데히드를 사용하여 고정하는 과정이 필요합니다.
고정과정을 거친 후 PI는 RNA에도 결합가능성이 있기에 RNase를 처리하여 염색시약이 오롯이 DNA에만 결합할 수 있도록 합니다.
세포주기 (Cell cycle) Protocol
- 세포배양
실험 목적에 알맞게 세포를 준비한 뒤 약물을 세포에 처리하여 반응합니다.
- 세포주기 sample 제작
1. 목적에 알맞게 준비한 세포를 PBS로 씻어줍니다.
2. 6well plate 기준으로 트립신을 처리하여 반응시킵니다.
3. 세럼이 포함된 미디어를 사용하여 세포를 15ml 튜브로 모아줍니다.
4. 원심분리를 수행합니다 (1500rpm, 5min).
5. 미디어를 폐기한 후 차가운 PBS로 pellet을 씻어주는 과정을 거칩니다.6. 차가운 70% 에탄올을 넣어 cell suspension을 시켜줍니다.
* 주의할 점은 세포가 뭉치지않게 약하게 vortexing을 하며 진행을 합니다.
→ 에탄올로 고정된 세포의 경우 4℃에서 몇 주 동안 안정적입니다.
→ 실험 스텝을 멈춰야 할 경우, 이 단계에서 세포를 저장합니다.
7. 원심분리를 수행합니다 (1500rpm, 5min).
8. 차가운 PBS로 pellet을 씻어주는 과정을 거칩니다.
9. 원심분리를 수행합니다 (1500rpm, 5min).
10. 각 샘플당 500ul 차가운 PBS로 cell suspension 과정을 거칩니다.11. 20mg/ml Rnase A 1.25ul 추가하여 실온에서 30분동안 반응시킵니다. incubation (RT)
12. 반응이 끝난 후 PI 염색시료를 샘플당 1mg/ml PI 15ul 추가해줍니다.
13. Flow cytometry 찍으시면 됩니다.'실험 프로토콜' 카테고리의 다른 글
IC50값 확인과 그래프 간단하게 그리기 (0) 2024.03.31 [Flow cytometry] 세포주기 (Cell cycle) Gating 및 분석 (0) 2024.03.27 약물 농도 계산 (몰 농도) 및 약물 처리하기 (0) 2024.03.27 Cell viability 측정 - WST-8 assay 원리 및 Protocol (0) 2024.03.22 Cell viability 측정 - MTT assay 원리 및 Protocol (0) 2024.03.22